Mahotas - Região de Filtragem
Neste artigo, veremos como podemos filtrar regiões em mahotas. Para isso, vamos usar a imagem de microscopia fluorescente de um benchmark de segmentação nuclear. Podemos obter a imagem com a ajuda do comando fornecido abaixo -
mhotas.demos.nuclear_image()
Abaixo está o nuclear_image
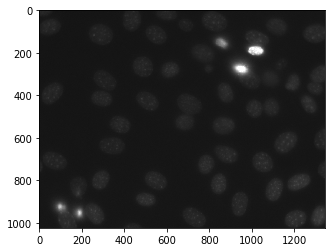
Para filtrar esta imagem vamos pegar o objeto de imagem que é numpy.ndarray e filtrá-lo com a ajuda de indexação, abaixo está o comando para fazer isso
nuclear = nuclear [:,:, 0]
Exemplo 1 :
importmahotas as mhimportmahotas.demosimportnumpy as npfrompylabimportimshow, shownuclear=mh.demos.nuclear_image()("Original Image i.e without filter")imshow(nuclear)show()nuclear=nuclear[:, :,0]("Image with filter")imshow(nuclear)show()
Resultado :

Exemplo 2:
importmahotas as mhimportmahotas.demosimportnumpy as npfrompylabimportimshow, shownuclear=mh.demos.nuclear_image()("Original Image i.e without filter")imshow(nuclear)show()nuclear=nuclear[500:,500:, :]("Image with filter")imshow(nuclear)show()
Resultado :

As postagens do blog Acervo Lima te ajudaram? Nos ajude a manter o blog no ar!
Faça uma doação para manter o blog funcionando.
70% das doações são no valor de R$ 5,00...

Diógenes Lima da Silva
