ML | Clustering espectral
Pré-requisitos: agrupamento de K-médias
Spectral Clustering é um algoritmo de clustering crescente que tem um desempenho melhor do que muitos algoritmos de clustering tradicionais em muitos casos. Ele trata cada ponto de dados como um nó de gráfico e, portanto, transforma o problema de agrupamento em um problema de particionamento de gráfico. Uma implementação típica consiste em três etapas fundamentais: -
- Construindo o Gráfico de Similaridade: Esta etapa constrói o Gráfico de Similaridade na forma de uma matriz de adjacência que é representada por A. A matriz de adjacência pode ser construída das seguintes maneiras: -
- Gráfico Epsilon-Vizinhança: Um parâmetro épsilon é fixado de antemão. Então, cada ponto é conectado a todos os pontos que estão em seu raio épsilon. Se todas as distâncias entre dois pontos forem semelhantes em escala, normalmente os pesos das arestas, ou seja, a distância entre os dois pontos, não são armazenados, uma vez que não fornecem nenhuma informação adicional. Assim, neste caso, o gráfico construído é um gráfico não direcionado e não ponderado.
- K-vizinhos mais próximos Um parâmetro k é fixado de antemão. Então, para dois vértices uev, uma aresta é direcionada de u para v apenas se v estiver entre os k-vizinhos mais próximos de u. Observe que isso leva à formação de um gráfico ponderado e direcionado porque nem sempre é o caso que para cada u tendo v como um dos k-vizinhos mais próximos, será o mesmo caso para v tendo u entre seus k-mais próximos vizinhos. Para tornar este gráfico não direcionado, uma das seguintes abordagens é seguida: -
- Direcione uma aresta de u para ve de v para u se v estiver entre os k-vizinhos mais próximos de u OU u estiver entre os k-vizinhos mais próximos de v.
- Direcione uma aresta de u para ve de v para u se v estiver entre os k-vizinhos mais próximos de u AND u estiver entre os k-vizinhos mais próximos de v.
- Gráfico Fully-Connected: para construir este gráfico, cada ponto é conectado com uma aresta não direcionada ponderada pela distância entre os dois pontos a cada outro ponto. Como essa abordagem é usada para modelar as relações de vizinhança locais, normalmente a métrica de similaridade gaussiana é usada para calcular a distância.
- Projetando os dados em um Espaço Dimensional inferior: Esta etapa é realizada para levar em conta a possibilidade de que membros do mesmo aglomerado possam estar distantes no espaço dimensional dado. Assim, o espaço dimensional é reduzido para que esses pontos fiquem mais próximos no espaço dimensional reduzido e, portanto, possam ser agrupados por um algoritmo de agrupamento tradicional. Isso é feito calculando a Matriz Laplaciana de Grafo . No entanto, para computá-lo primeiro, o grau de um nó precisa ser definido. O grau do iº nó é dado por
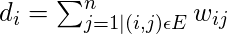
Observe que
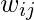 é a aresta entre os nós i e j conforme definido na matriz de adjacência acima.
é a aresta entre os nós i e j conforme definido na matriz de adjacência acima.A matriz de grau é definida da seguinte forma: -
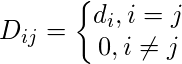
Assim, o gráfico Laplacian Matrix é definido como: -
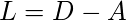
Essa matriz é então normalizada para eficiência matemática. Para reduzir as dimensões, primeiro são calculados os autovalores e os respectivos autovetores. Se o número de clusters é k, então os primeiros autovalores e seus autovetores são tomados e empilhados em uma matriz de modo que os autovetores sejam as colunas.
- Clustering the Data: Este processo envolve principalmente o clustering dos dados reduzidos usando qualquer técnica tradicional de clustering - normalmente K-Means Clustering. Em primeiro lugar, cada nó é atribuído a uma linha do normalizado da Matriz Laplaciana do gráfico. Em seguida, esses dados são agrupados usando qualquer técnica tradicional. Para transformar o resultado do armazenamento em cluster, o identificador de nó é retido.
Propriedades:
- Suposição-menos: esta técnica de agrupamento, ao contrário de outras técnicas tradicionais, não assume que os dados seguem alguma propriedade. Portanto, isso faz com que essa técnica responda a uma classe mais genérica de problemas de agrupamento.
- Facilidade de implementação e velocidade: Este algoritmo é mais fácil de implementar do que outros algoritmos de agrupamento e também é muito rápido, pois consiste principalmente em cálculos matemáticos.
- Não escalonável: uma vez que envolve a construção de matrizes e cálculo de autovalores e autovetores, é demorado para conjuntos de dados densos.
As etapas abaixo demonstram como implementar Spectral Clustering usando Sklearn. Os dados para as etapas a seguir são os dados do cartão de crédito, que podem ser baixados do Kaggle .
Etapa 1: Importar as bibliotecas necessárias
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.cluster import SpectralClustering
from sklearn.preprocessing import StandardScaler, normalize
from sklearn.decomposition import PCA
from sklearn.metrics import silhouette_scoreEtapa 2: Carregando e limpando os dados
# Changing the working location to the location of the data
cd "C:\Users\Dev\Desktop\Kaggle\Credit_Card"
# Loading the data
X = pd.read_csv('CC_GENERAL.csv')
# Dropping the CUST_ID column from the data
X = X.drop('CUST_ID', axis = 1)
# Handling the missing values if any
X.fillna(method ='ffill', inplace = True)
X.head()
Etapa 3: pré-processamento dos dados para torná-los visualizáveis
# Preprocessing the data to make it visualizable
# Scaling the Data
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
# Normalizing the Data
X_normalized = normalize(X_scaled)
# Converting the numpy array into a pandas DataFrame
X_normalized = pd.DataFrame(X_normalized)
# Reducing the dimensions of the data
pca = PCA(n_components = 2)
X_principal = pca.fit_transform(X_normalized)
X_principal = pd.DataFrame(X_principal)
X_principal.columns = ['P1', 'P2']
X_principal.head()
Etapa 4: Construindo os modelos de clustering e visualizando o clustering
Nas etapas a seguir, dois modelos diferentes de Spectral Clustering com valores diferentes para o parâmetro 'afinidade'. Você pode ler sobre a documentação da classe Spectral Clustering aqui .
a) afinidade = 'rbf'
# Building the clustering model
spectral_model_rbf = SpectralClustering(n_clusters = 2, affinity ='rbf')
# Training the model and Storing the predicted cluster labels
labels_rbf = spectral_model_rbf.fit_predict(X_principal)# Building the label to colour mapping
colours = {}
colours[0] = 'b'
colours[1] = 'y'
# Building the colour vector for each data point
cvec = [colours[label] for label in labels_rbf]
# Plotting the clustered scatter plot
b = plt.scatter(X_principal['P1'], X_principal['P2'], color ='b');
y = plt.scatter(X_principal['P1'], X_principal['P2'], color ='y');
plt.figure(figsize =(9, 9))
plt.scatter(X_principal['P1'], X_principal['P2'], c = cvec)
plt.legend((b, y), ('Label 0', 'Label 1'))
plt.show()
b) afinidade = 'mais próximos_ vizinhos'
# Building the clustering model
spectral_model_nn = SpectralClustering(n_clusters = 2, affinity ='nearest_neighbors')
# Training the model and Storing the predicted cluster labels
labels_nn = spectral_model_nn.fit_predict(X_principal)
Etapa 5: Avaliação do desempenho
# List of different values of affinity
affinity = ['rbf', 'nearest-neighbours']
# List of Silhouette Scores
s_scores = []
# Evaluating the performance
s_scores.append(silhouette_score(X, labels_rbf))
s_scores.append(silhouette_score(X, labels_nn))
print(s_scores)
Etapa 6: Comparando os desempenhos
# Plotting a Bar Graph to compare the models
plt.bar(affinity, s_scores)
plt.xlabel('Affinity')
plt.ylabel('Silhouette Score')
plt.title('Comparison of different Clustering Models')
plt.show()
As postagens do blog Acervo Lima te ajudaram? Nos ajude a manter o blog no ar!
Faça uma doação para manter o blog funcionando.
70% das doações são no valor de R$ 5,00...

Diógenes Lima da Silva
